Con il crescente utilizzo delle tecniche di Machine Learning (ML) in varie industry, stiamo assistendo ad un aumento del numero di progetti e della loro complessità. Questo genera da un lato l’esigenza di maggiore governance ovvero capacità di orchestrare e controllare la catena di sviluppo e rilascio sull’intera ciclo di vita del ML (preprocessing, model training, testing, deployment) e dall’altro lato l’esigenza di scalabilità, ovvero riuscire a replicare in maniera efficiente parti intere del processo per gestire molteplici modelli di ML (Machine Learning).
Una recente ricerca in USA, effettuata per comprendere le tendenze relative al mondo del Machine Learning per il 2021, ha elaborato un sondaggio effettuato su un campione significativo di 400 aziende di varia dimensione: il 50% di queste sta gestendo attualmente più di 25 modelli di ML e il 40% del totale gestisce oltre 50 modelli di ML. Tra le organizzazioni di grande dimensione (oltre 25.000 collaboratori) il 41% di esse possiede oltre 100 algoritmi di ML in produzione!
La sfida per queste organizzazioni negli anni avvenire sarà quella di riuscire a gestire questa crescita in modo da capitalizzare il know-how e poter manutenere e controllare tutti questi modelli riducendo la complessità. In altre parole, riuscire a gestire una crescita esplosiva di applicazioni per Machine Learning senza dover aumentare il livello di complessità degli stesso ordine di grandezza.
E’ proprio qui che intervengono le Best Practice di MLOps, letteralmente Machine Learning Operations: strumenti e procedure finalizzate alla gestione della catena di processi di ML per ottimizzarne la governance e la scalabilità.
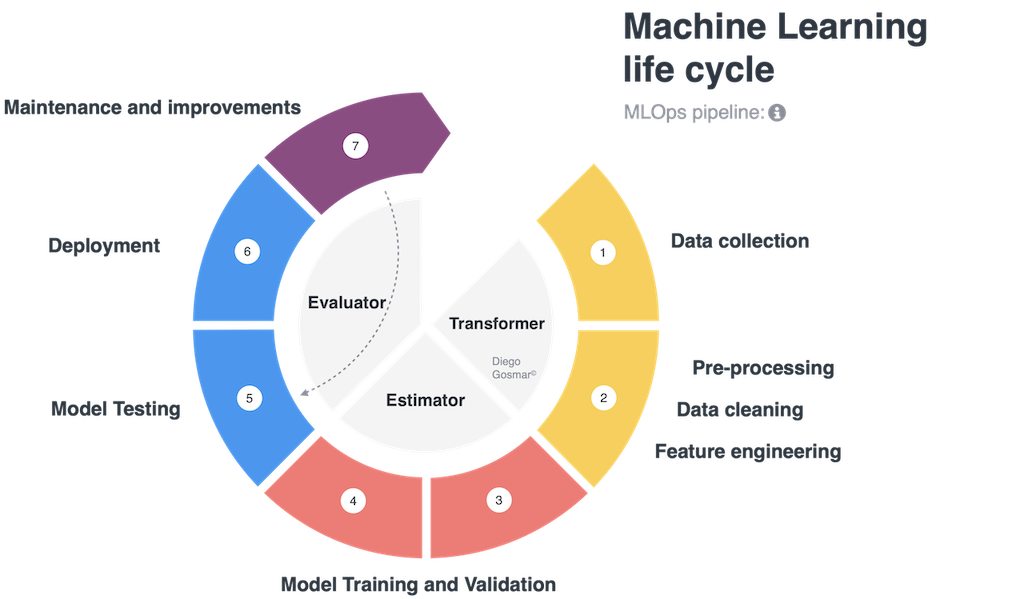
Uno dei metodi più recenti per indirizzare controllo e scalabilità con ML è l’utilizzo di pipeline. Un ciclo di design, training, test e rilascio in produzione per affrontare una applicazione di intelligenza artificiale con Machine Learning è tipicamente costituito dalle fasi seguenti: data collection, data cleaning, feature engineering (i.e. scaling, normalizing, labelling, dimensionality reduction, binning, encoding etc…), Model Training and Validation,
Model Testing, Model deploying, Continuous improvement. Parliamo anche di ML life cycle per identificare la catena di step che coinvolgono questi processi.
E’ interessante notare come molti di questi step possano essere riutilizzati in differenti contesti su molteplici applicazioni di AI. Ad esempio, se desideriamo testare differenti algoritmi di machine learning (siano ad esempio 3 modelli differenti basati su Decision Tree, Deep Learning Neural Network e SVM) sullo stesso dataset, i primi step di data collection, data cleaning, feature engineering saranno analoghi per ognuno degli algoritmi che desidero testare, mentre la fase di model training, validation and test cambierà: le ML pipeline indirizzano esattamente questi scenari rendendo la il life cycle modulare, controllabile, ripetibile… in definitiva scalabile!
Vediamo ora un caso pratico di utilizzo delle pipeline per applicazioni di MLOps considerando un esempio semplice di regressione lineare e utilizzando il framework PySpark messo a disposizione da Databricks.
PySpark è un linguaggio di derivazione Python che eredita i benefici di Apache Spark, progettato per eseguire analisi e pre-processing di dataset su larga scala e costruire pipeline per machine learning al fine di rendere l’intero processo estremamente scalabile… Il tutto su architettura distribuita per ottimizzare la scalabilità.
Grazie al concetto pipeline andremo a gestire un flusso di lavoro eseguibile attraverso moduli indipendenti che andranno a comporre l’attività di Machine Learning completa. I moduli sono gestiti come una serie di passaggi all’interno della pipeline che interagiscono tra di loro tramite delle API
Se ci riflettiamo bene, questo tipo di soluzione è analoga al paradigma di gestione delle applicazioni su cloud attraverso micro-servizi: per chi conosce le applicazioni offerte su container da brand più o meno blasonati, l’idea è fondamentalmente quella di gestire problematiche complesse andando a “spezzettarle” in contenitori più semplici possibili, ognuno dei quali indipendente, facilmente replicabile e sostituibile al bisogno.
Il tutto risulta, inoltre, molto più efficiente quando ci troviamo di fronte alla gestione di applicazioni di AI, che necessitino di orchestrare machine learning life cycle a “diverse mani”, ovvero da un team di data scientist eterogeneo e numeroso: utilizzando l’approccio modulare delle pipeline, possiamo garantire accessi indipendenti (quindi maggiore sicurezza) oltre a mantenere test e provisioning estremamente più sotto controllo.
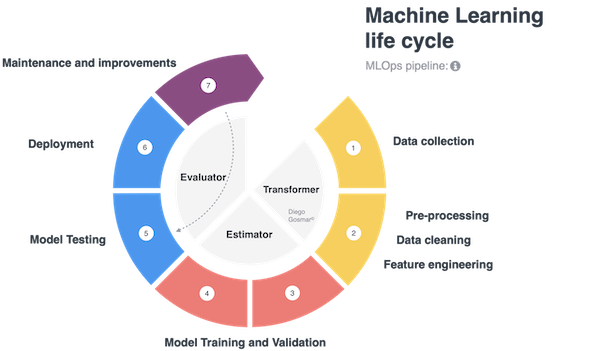
Una pipeline è costituita tipicamente dai tre elementi seguenti:
Trasformatore: un algoritmo che può trasformare un Dataset in un altro Dataset. Ad esempio, un modello addestrato di ML è un trasformatore, poichè trasforma un DataFrame con feature indipendenti in un DataFrame con le label valorizzate con le loro predizioni. Anche le fasi di preprocessing per elaborare il dataset prima di darlo in pasto alle fasi di training vengono gestite da trasformatori.
Stimatore: un algoritmo che viene addestrato su un Dataset per produrre un Trasformatore. Ad esempio, un modello di apprendimento di ML (fase di training) è uno stimatore che esegue il training su un DataFrame e produce un “modello addestrato” (che andremo poi a testare e utilizzare per effettuare le predizioni).
In altre parole: parleremo di stimatori durante i processi di Training e di trasformatori durante i processi di pre-processing (feature engineering etc…) e quelli di Predizione (o inferenza).
Infine useremo degli Evaluator per analizzare accuratezza e bontà del modello addestrato: gli evaluator o Valutatori saranno pertanto utilizzati durante le fasi di Testing.
Torniamo al nostro esempio con Pyspark.
Come accennato in precedenza, grazie a Databricks e Spark possiamo programmare ed eseguire il nostro ciclo di machine learning su un’architettura distribuita (cluster di nodi che possono lavorare in parallelo), beneficiando quindi di un ulteriore importante livello di scalabilità.

Una volta creato un cluster adeguato su Databricks possiamo uploadare i nostri dati. Lo facciamo inizialmente in formato nativo .csv, ma poi andiamo a convertire le nostre tabelle in formato colonnare Apache Parquet: questo ci consente di sfruttare al meglio la modalità lazy execution di Spark.
Con la modalità eager execution, ogni operazione del ciclo di una pipeline viene eseguita il più rapidamente possibile, portando tutti i dati in memoria. La modalità lazy execution lavora invece andando a portare in memoria i dati solo nel momento in cui sono davvero indispensabili a completare l’operazione. Questo si traduce in un processo ottimizzato per lavorare su architetture distribuite come Databricks o Hadoop.
Per eseguire le nostre operazioni, Databricks mette a disposizione un comodissimo ambiente di lavoro basato su Notebook (del tutto simile a quello che potete usare con Anaconda e Jupyter Notebook, oppure Google Colab, Amazon Sagemaker e numerosi altri ambienti…).
La prima cosa che dobbiamo fare è “agganciare” il nostro Notebook al Cluster di riferimento.
Quindi iniziamo con importare il nostro dataset in formato .csv, dopo averlo opportunamente caricato sul datastore del cluster:
file_location = "/FileStore/tables/ais-4.csv"
df = spark.read.format("csv").option("inferSchema",
True).option("header", True).load(file_location)
Visualizziamone lo schema:
df.printSchema()
(1) Spark Jobsdf:pyspark.sql.dataframe.DataFrame = [_c0: integer, rcc: double … 12 more fields]root |– _c0: integer (nullable = true) |– rcc: double (nullable = true) |– wcc: double (nullable = true) |– hc: double (nullable = true) |– hg: double (nullable = true) |– ferr: integer (nullable = true) |– bmi: double (nullable = true) |– ssf: double (nullable = true) |– pcBfat: double (nullable = true) |– lbm: double (nullable = true) |– ht: double (nullable = true) |– wt: double (nullable = true) |– sex: string (nullable = true) |– sport: string (nullable = true)
Si tratta del dataset relativo ai parametri di esami del sangue degli atleti australiani, che abbiamo già utilizzato in uno dei nostri esempi precedenti di linear regression!
Convertiamo il dataset da csv a parquet, per lavorare in modalità lazy execution, come già illustrato (questo sarà da eseguire solo la prima volta):
df.write.save('/FileStore/parquet/ais-4',format='parquet')
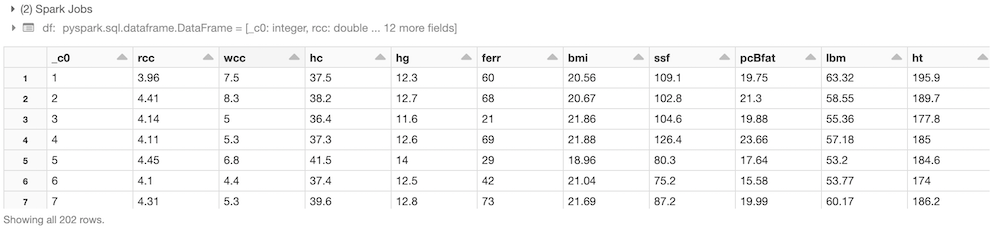
Carichiamo i dati in formato parquet e diamo un’occhiata al nostro dataset!df = spark.read.load("/FileStore/parquet/ais-4")
display(df)
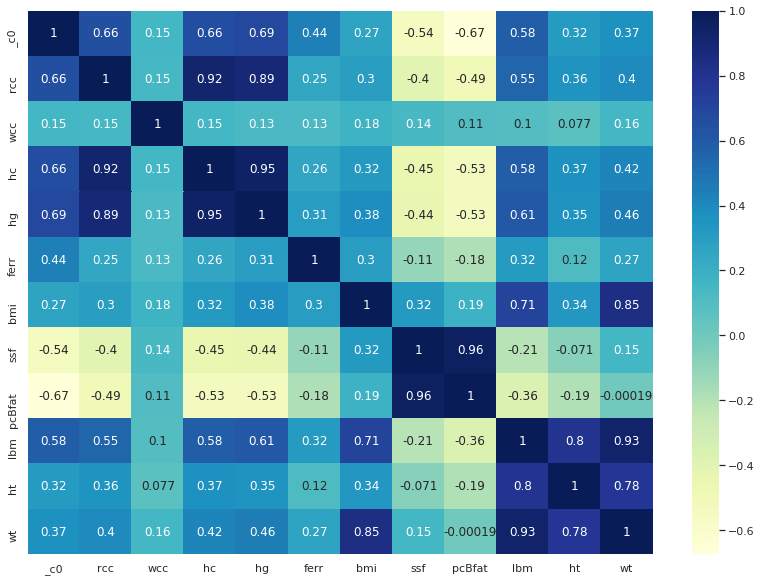
Valutiamo la correlazione delle feature del nostro dataset ed effettuiamo opportune riduzioni dimensionali:
import matplotlib.pyplot as plt
import seaborn as sns; sns.set()
df2 = df.toPandas()
fig, ax = plt.subplots(figsize=(14,10))
ax = sns.heatmap(df2.corr(), cmap="YlGnBu", annot = True)
df_new=df.drop('rcc','hc','hg','ferr','ssf','pcBfat','sport')
df_new.printSchema()
Splittiamo il dataset per sesso maschile e femminile:
df_male = df_new.filter(df_new.sex == "m")
df_female = df_new.filter(df_new.sex == "f")
Predisponiamo l’ambiente per utilizzare le pipeline e le librerie MLib: un set molto potente specifico per implementare modelli di machine learning in ambienti distribuiti come Spark!
from pyspark.ml.feature import VectorAssembler
from pyspark.ml.regression import LinearRegression
from pyspark.ml import Pipeline
Le MLib prevedono che le feature indipendenti siano passate come unico vettore e la label da predire come valore scalare:
#set the independent variable vector (features)
assembler = VectorAssembler(inputCols=['bmi', 'wt'],outputCol="features")
Utilizziamo quindi la funzione LinearRegression messa a disposizione dal MLib, nel nostro caso per predire il valore di lbm dell’atleta (Lean Body Mass ovvero Indice di Massa Magra):
lr = LinearRegression(featuresCol = 'features', labelCol='lbm')
Separiamo i dati per la fase di Training (80% dei dati) e per quella di Test (20% dei dati):
train_df, test_df = train.randomSplit([0.8, 0.2]) ;to be used in case of no Pipeline commands
train_df, test_df = df_male.randomSplit([0.8, 0.2])
A questo punto è venuto il momento di mettere insieme il ciclo di ML utilizzando la funzione pipeline: nel nostro caso abbiamo una fase di preprocessing, identificata con la risultante della variabile assembler che racchiude le feature engineering cha abbiamo eseguito in precedenza e quella dell’estimatore, la variabile lr che implementa il modello di regressione lineare nel nostro caso:
pipeline = Pipeline(stages=[assembler, lr])
Utilizziamo il metodo fit della pipeline per eseguire quindi l’addestramento del modello:lr_model = pipeline.fit(train_df)
Una volta effettuato il training e validation, andiamo a testare il nostro modello. Per farlo invochiamo il metodo transform della nostra pipeline, che utilizzeremo per applicare il modello addestrato (lr_model nel nostro caso) ai dati di test:
predictions = lr_model.transform(test_df)
predictions.show()
Ecco il risultato delle predizioni effettuate sul dataset di test:
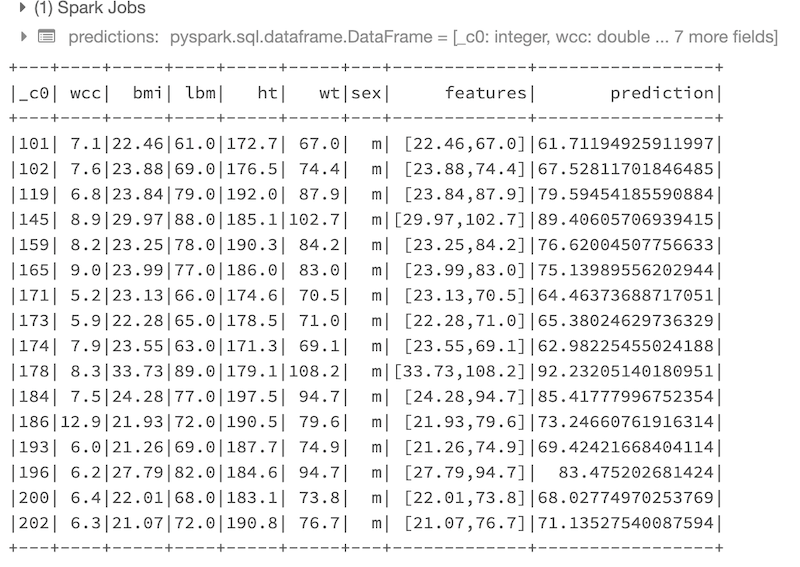
A questo punto utilizziamo opportune funzioni “Evaluator” per visualizzare le metriche ovvero i risultati più rilevanti del modello di regressione addestrato (sempre sulle predizioni effettuate sul dati di test):
from pyspark.ml.evaluation import *
eval_rmse = RegressionEvaluator(labelCol="lbm", predictionCol="prediction", metricName="rmse")
eval_mse = RegressionEvaluator(labelCol="lbm", predictionCol="prediction", metricName="mse")
eval_mae = RegressionEvaluator(labelCol="lbm", predictionCol="prediction", metricName="mae")
eval_r2 = RegressionEvaluator(labelCol="lbm", predictionCol="prediction", metricName="r2")
print('RMSE:', eval_rmse.evaluate(predictions))
print('MSE:', eval_mse.evaluate(predictions))
print('MAE:', eval_mae.evaluate(predictions))
print('R2:', eval_r2.evaluate(predictions))
RMSE e R2 sono i due parametri più significativi per valutare l’accuratezza del nostro modello di regressione. Ecco i risultati:
(4) Spark Jobs
RMSE: 2.4854195595721813
MSE: 6.177310387103976
MAE: 1.5654423776210198
R2: 0.907450608117363
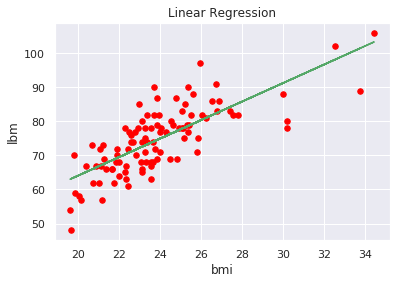
Possiamo anche visualizzare l’andamento della nostra funzione di regressione per avere un’idea grafica del risultato (semplifichiamolo selezionando solo una variabile indipendentemente bmi per avere un plot bidimensionale):
import numpy as np
from numpy import polyfit
data = df_male
x1 = data.toPandas()['bmi'].values.tolist()
y1 = data.toPandas()['lbm'].values.tolist()
plt.scatter(x1, y1, color='red', s=30)
plt.xlabel('bmi')
plt.ylabel('lbm')
plt.title('Linear Regression')
p1 = polyfit(x1, y1, 1)
plt.plot(x1, np.polyval(p1,x1), 'g-' )
plt.show()
Abbiamo visto un esempio su come adottare alcune best practice di pipeline su ambienti distribuiti con Spark per orchestrare in maniera scalabile i nostri processi di machine learning.
Esistono ulteriori tool che consentono di indirizzare l’assessment delle pipeline e gestire le MLOps in maniera professionale per migliorare la governance e la scalabilità: stay tuned!
Volete approfondire meglio questi argomenti?
Ecco una lettura interessante su Machine Learning e AI.